C++的作者B.Stroustrup 说过世界上只有两种语言,一种经常有人对它抱怨,还有一种根本没人用(比如___,免得我被打请大家自行填空_(┐「ε:)_ )。
Python最经常被人抱怨的就是慢(大概比c要慢40倍,的确是挺感人的…具体数据可以参考这里)和它的gil(没有真正的线程这一点是经常遭到诟病的地方)。的确,现在cpu已经很快很快了,对于Python这样一门表达能力超强的语言,我们在一定程度上可以对它的速度有所忍受,并且对于网络应用来说python通过异步来实现并发也是可以非常快的。但Python 有时也会被用来做一些计算密集型的工作,这个时候就需要一些办法来加速了,现在看来有一大票办法来加速Python,并且其中有一些能够一定程度上解决gil的问题,最近我学习了其中几种,这里就结合一个例子来简单谈一下感受。
一个计算密集型过程例子:julia set
julia set 与 mandelbrot set 一样,是一种分形结构,它能由极其简单的规则所描述,但产生出的图形却十分的迷人与复杂。julia set的计算规则真的非常简单,以便理解后面的代码我先说明一下,已经理解的朋友请跳过这一部分。
julia set可以由

现在我们来尝试用python来计算出一个julia set的图形,并用几种方法对这个过程进行加速,来看看它们的加速效果。
pure Python
首先我们用纯Python实现julia set的计算,为对比后面各种手段的加速效果提供参考。
# jl.py
# calc_julia pure python edition
import numpy as np
def escape(z, c, z_max, n_max):
i = 0
while (z.real**2 + z.imag**2 < z_max**2) and i < n_max:
z = z*z + c
i += 1
return i
def calc_julia(resolution, c, bound=1.5, z_max=4, n_max=1000):
step = 2.0 * bound / resolution
counts = np.zeros((resolution + 1, resolution + 1), dtype=np.int32)
for i in range(resolution + 1):
real = -bound + i * step
for j in range(resolution + 1):
imag = -bound + j * step
z = real + imag * 1j
counts[i, j] = escape(z, c, z_max, n_max)
return np.asarray(counts)
注意这里虽然import了numpy但是并没有使用numpy进行计算,只是把计算结果存储在一个numpy数组里方便以使用matplotlib进行绘图。然后我们可以使用calc_julia这个函数计算得到julia set并对它进行可视化了。 打开ipython notebook:
In [1]: import matplotlib.pyplot as plt
In [2]: import numpy as np
In [3]: from jl import calc_julia
In [4]: jls = calc_julia(1000, -0.4+0.6j)
In [5]: plt.imshow(np.log(jls));plt.show()
这样我们就会得到对于复数 -0.4 + 0.6i 的julia set 图形,如下,感觉挺神奇的╰( ᐖ╰)。
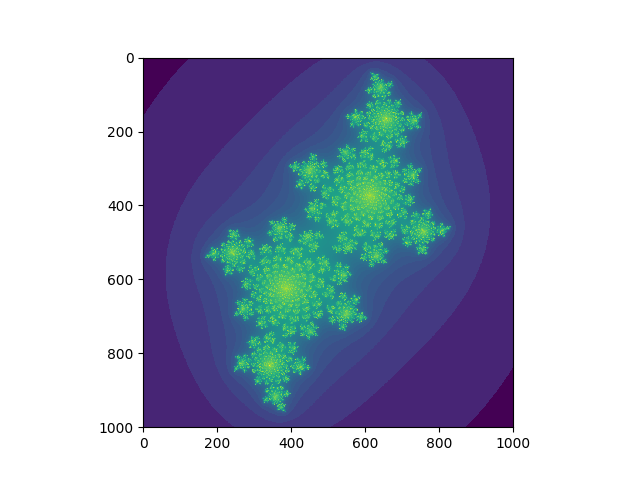
这很有趣但是计算jls这一步的确是慢了一点,大概要7.6秒,如果我们想多看几个其他复数的julia set,恐怕是等的会有点烦了。我们下面来看一看加速后这个过程要多久吧。
numba
numba大概是使用起来最简单的计算加速办法了吧,只需要在函数上面加一个小小的装饰器,numba就会把这个函数编译成LLVM的IR码从而达到jit的加速效果。我们来试一下。
# jl.py
# calc_julia numba edition
import numpy as np
from numba import jit
@jit
def escape(z, c, z_max, n_max):
i = 0
while (z.real**2 + z.imag**2 < z_max**2) and i < n_max:
z = z*z + c
i += 1
return i
@jit
def calc_julia(resolution, c, bound=1.5, z_max=4, n_max=1000):
step = 2.0 * bound / resolution
counts = np.zeros((resolution + 1, resolution + 1), dtype=np.int32)
for i in range(resolution + 1):
real = -bound + i * step
for j in range(resolution + 1):
imag = -bound + j * step
z = real + imag * 1j
counts[i, j] = escape(z, c, z_max, n_max)
return np.asarray(counts)
看很简单吧,只需要在函数上面加上@jit这个装饰器即可。测试一下速度,完成与上面相同的计算只需要0.6s比纯python快了12.6倍左右。
PyPy
pypy是一个jit的Python解释器实现,不需要修改代码就能执行,也相对比较方便,但是毕竟它已经脱离了CPython的生态,配置起来比起numba那样直接一条pip指令就能安装还是稍微麻烦一点,并且还有好多Cython中的库在这里也是没有的。把上面纯Python的代码用PyPy运行一遍,耗时0.44s,效果很可观,甚至比numba还要快一点。
Cython
Cython即带有C类型的Python,通过引入C类型把Cython代码编译成高效的C代码,相当于使用类型信息对Python代码的“蒸馏”,并且“蒸馏”的粒度是可控的,纯Python代码在Cython中完全是合法的,只是引入越多的类型信息编译出来的代码就越高效,并且还能用来方便地包装C代码和使用C库,可以说Cython是这些方案之中最强大最成熟的一种,很多大型项目诸如Scipy、Pandas、sklearn中都大量使用了Cython。它用来写库是一种非常好的选择,对于Python用户来说Cython是非常值得学习的,如果感兴趣的话,有一本很好的讲Cython的书Cython A Guide for Python Programmers可以找来阅读一下,网上很容易找到电子版。以下的计算julia set的Cython代码就来源于这本书。
# jl.pyx
# calc_julia cython edition
from cython import boundscheck, wraparound
import numpy as np
cdef int escape(double complex z,
double complex c,
double z_max,
int n_max) nogil:
cdef:
int i = 0
double z_max2 = z_max * z_max
while norm2(z) < z_max2 and i < n_max:
z = z * z + c
i += 1
return i
cdef inline double norm2(double complex z) nogil:
return z.real * z.real + z.imag * z.imag
@boundscheck(False)
@wraparound(False)
def calc_julia(int resolution, double complex c,
double bound=1.5, double z_max=4.0, int n_max=1000):
cdef:
double step = 2.0 * bound / resolution
int i, j
double complex z
double real, imag
int[:, ::1] counts
counts = np.zeros((resolution + 1, resolution + 1), dtype=np.int32)
for i in range(resolution + 1):
real = -bound + i * step
for j in range(resolution + 1):
imag = -bound + j * step
z = real + imag * 1j
counts[i, j] = escape(z, c, z_max, n_max)
return np.asarray(counts)
恩比起numba与pypy似乎麻烦了一点,多了很多以cdef开头的类型声明,不过当你掌握了它简单的语法以后会发现这并不难就像Python一样。编写完.pyx文件后与之前不同的是这里需要编译Cython代码,所以我们再创建一个名为setup.py的文件。
# setup.py
from distutils.core import setup
from Cython.Build import cythonize
setup(name="julia",
ext_modules=cythonize("jl.pyx"))
然后在终端内敲入python setup.py build_ext -i进行编译,然后就能与之前一样的方式使用编译好的函数了,运行一下,计算过程耗时0.16s,比pypy更快了,比最初的纯Python版本快了40多倍!
结语
这里说了三种加速Python的办法,总结一下就是写独立的应用(不需要太多的第三方库的)可以试试PyPy,加速函数运行速度就用numba,写库就用Cython。其实还有很多加速方案啦,比如使用将计算向量化使用numpy,以及它的多线程版numexper,以及使用多进程multiprocessing多进程编程等等,具体选哪种还要看情况,当然对于优化速度来说最重要的还是选对合适的算法。并且在优化之前要想清楚现阶段需不需要优化,很多时候对于比如科学计算这样的应用,比起速度可能更在意的还是正确性,先把功能做对了再来看优化吧。
Email: nanguage@yahoo.com

 (C:)
(C:) 3d
3d chain-thought
chain-thought
 web-python-console
web-python-console
 SRT data analysis
SRT data analysis
 JupyterLite
JupyterLite
 U-FISH
U-FISH
 写给生物信息初学者
写给生物信息初学者
 AniNet
AniNet
 Boids
Boids
 EM 算法 1. how it works
EM 算法 1. how it works
 翻译:Inside Python Virtual Machine(前三章)
翻译:Inside Python Virtual Machine(前三章)
 PubMed bio-conception network example
PubMed bio-conception network example
 three.js test page
three.js test page
